Kit de ensayo de quinasa fluorimétrica universal Amplite® *Fluorescencia roja* Este kit proporciona un ensayo rápido, simple y homogéneo para medir la actividad de las quinasas.
Descripción
Kit de ensayo de quinasa fluorimétrico universal Amplite® *Fluorescencia roja*
La mayoría de los kits comerciales de ensayo de proteína quinasa se basan en el control de la formación de fosfopéptidos o en la eliminación de ATP. Para los kits de ensayo de quinasa que se basan en la detección de fosfopéptidos, se debe dedicar tiempo y esfuerzo para identificar un sustrato peptídico optimizado, mientras que el método de depleción de ATP sufre varias interferencias debido al uso de luciferasa que son inhibidas o activadas por varios compuestos biológicos. El kit de ensayo de cinasa universal Amplite® se basa en el control de la formación de ADP, que es directamente proporcional a la actividad de la enzima fosfotransferasa y se mide fluorométricamente.
Este kit proporciona un ensayo rápido, simple y homogéneo para medir la actividad de las quinasas. Las características de su alta sensibilidad (<0,2 uM ADP), amplia tolerancia ATP (1-300 uM), método no basado en anticuerpos, no radiactivo y sin lavado para detectar la cantidad de ADP producido como resultado de la actividad enzimática hacen un kit ideal para determinar la cinética de la cinasa Michaelis-Menten y para detectar e identificar inhibidores de la cinasa. El ensayo se puede realizar en un conveniente formato de placa de microtitulación de 96 pozos o de 384 pozos y se puede adaptar fácilmente a la automatización sin necesidad de pasos de separación.
| Catalogo | Producto | Presentación |
|---|---|---|
| AAT-31001 | Kit de ensayo de quinasa fluorimétrico universal Amplite® *Fluorescencia roja* | 250 pruebas |
| AAT-31002 | Kit de ensayo de quinasa fluorimétrico universal Amplite® *Fluorescencia roja* | 500 pruebas |
Importante: Solo para uso en investigación (RUO).
Plataforma
Lector de microplacas de Fluorescencia
| Excitación | 540 nm |
| Emisión | 590 nm |
| Cutoff | 570 nm |
| Placa recomendada | Negra sólida |
Componentes
| Componente A: ADP Sensor Buffer | 1 vial (5 mL) |
| Componente B1: ADP Sensor I (Sensible a la luz) | 1 vial (en polvo) |
| Componente B2: ADP Sensor II | 1 vial (2.5 mL) |
| Componente B3: DMSO | 1 vial (100 µL) |
| Componente C: ADP Standard | 1 vial |
| Componente D: ADP Assay Buffer | 1 vial (10 mL) |
PREPARACION DE SOLUCION DE STOCK
A menos que se indique lo contrario, todas las soluciones madre no utilizadas deben dividirse en alícuotas de un solo uso y almacenarse a -20 °C después de la preparación. Evite los ciclos repetidos de congelación y descongelación.
- Solución madre del sensor I de ADP (50X):
Agregue 50 µL de DMSO (Componente B3) en el vial del Sensor 1 de ADP (Componente B1) para preparar una solución madre del Sensor I de ADP 50X. - Solución estándar de ADP (300 mM):
Agregue 100 µl de ddH2O al estándar de ADP (componente C) para hacer una solución estándar de ADP de 300 mM.
PREPARACION DE SOLUCION ESTANDAR
Estándar ADP
Para mayor comodidad, utilice el Planificador de dilución en serie: https://www.aatbio.com/tools/serial-dilution/31001
Tome una solución estándar de ADP de 300 mM y diluya 10.000X en buffer de reacción de quinasa para hacer una solución estándar de ADP de 30 µM. Tome una solución estándar de ADP de 30 µM y realice una dilución en serie 1:3 en buffer de reacción de quinasa para diluir en serie la solución estándar de ADP. Nota: Realice diluciones en serie del estándar de ADP en el buffer de reacción de quinasa incluyendo una muestra sin ADP para medir la fluorescencia de fondo.
PREPARACION DE SOLUCION DE TRABAJO
Agregue 50 µl de solución madre del sensor I de ADP 50X en el vial del sensor II de ADP (componente B2) para que la solución del sensor de ADP funcione. Nota: El sensor ADP reconstituido no es estable, renueve según sea necesario.
Imagenes
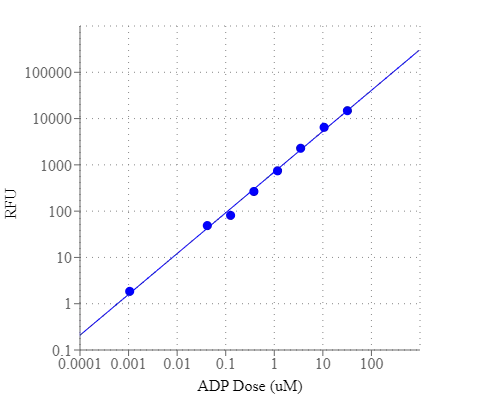
Figura 1. La respuesta a la dosis de ADP se midió con el kit de ensayo de quinasa fluorimétrica universal Amplite® en una placa negra sólida de 384 pozos utilizando un lector de microplacas de fluorescencia Gemini (Molecular Devices).
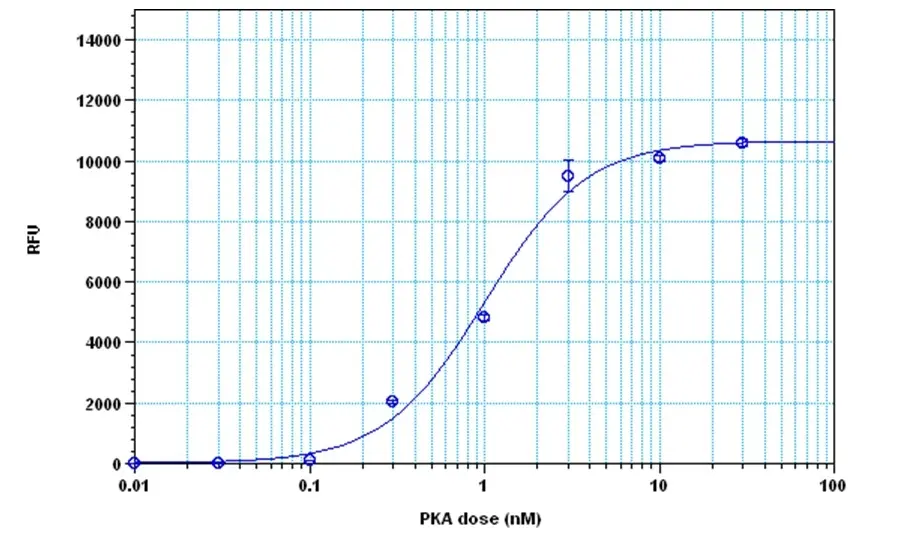
Figura 2. Detección de proteína quinasa A con el kit de ensayo de quinasa fluorimétrica Amplite®. La quinasa se incubó en presencia de ATP y sustrato peptídico de kemptide durante 30 minutos, y la generación de ADP se detectó después de 30 minutos de incubación utilizando el kit de ensayo de quinasa fluorimétrica Amplite®.
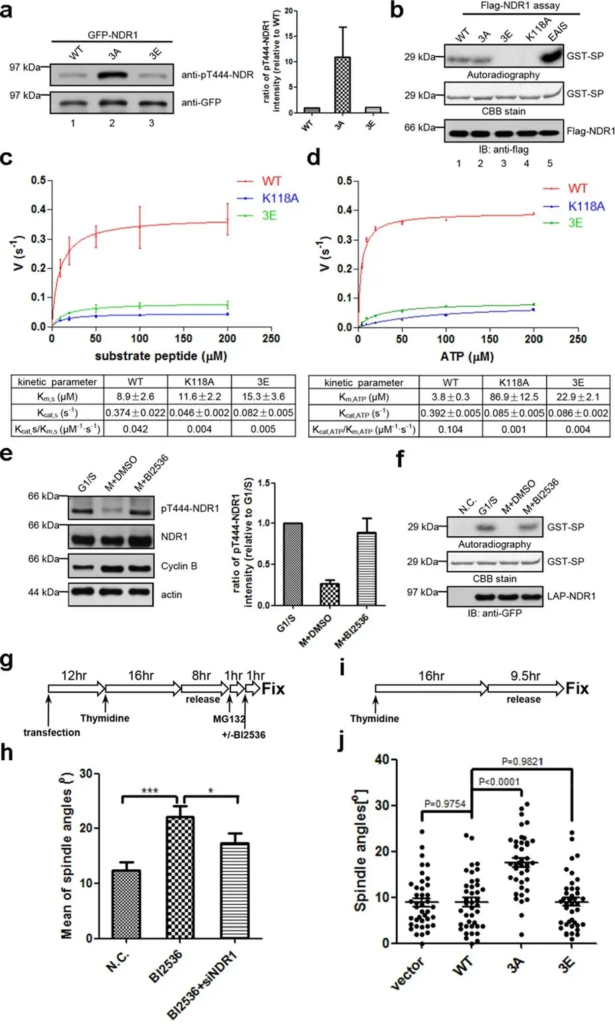
Figura 3. La fosforilación mediada por PLK1 suprimió la actividad de NDR1 para garantizar la orientación adecuada del huso. (a) Inmunotransferencia de lisados de células HeLa mitóticas que expresan GFP-NDR1WT, GFP-NDR13A o GFP-NDR13E, con anticuerpos anti-GFP y anti-pT444-NDR1. La cuantificación se muestra a la derecha. Las barras representan medias ± SD, n = 3. (b) Medida de la actividad de la cinasa de Flag-NDR1 de tipo salvaje o mutantes (3A, 3E, K118A y EAIS) purificada a partir del ensayo de cinasa in vitro de células 293 T asíncronas. Las transferencias/geles completos se presentan en la figura complementaria S4. (c) y (d) Determinación de los parámetros cinéticos de NDR1WT, NDR1K118A y NDR13E. Las velocidades del ensayo de cinasa hacia el péptido sustrato de 10 mer (c) o ATP (d) a concentraciones variables se midieron con el kit de ensayo de cinasa fluorimétrica universal AmpliteTM. Los datos de tres experimentos independientes se analizaron en GraphPad Prism y se ajustaron con la ecuación de Michaelis-Menten para extraer los parámetros cinéticos. Las barras indican la media ± DE. (e) Análisis de inmunotransferencia de lisados de células HeLa sincronizados con tratamiento (+BI2536) o sin (+DMSO) BI2536, con los anticuerpos contra las proteínas indicadas. “M” representa mitosis. La cuantificación se muestra a la derecha. Las barras representan medias ± SD de 3 experimentos independientes. Las transferencias/geles de longitud completa se presentan en la Figura complementaria S4. (f) Medida de la actividad de la cinasa de LAP-NDR1 purificada a partir de células HeLa de expresión estable LAP-NDR1 sincronizadas con tratamiento (+BI2536) o sin (+DMSO) BI2536, o HeLa normal células (N.C.) ensayo de quinasa in vitro. Las transferencias / geles de longitud completa se presentan en la Figura complementaria S4. ( g ) Descripción general esquemática del procedimiento experimental de ( h ). ( h ) Cuantificación de los ángulos del huso de las células HeLa mitóticas que expresan siRNA NDR1 (+siNDR1) o no, con o sin tratamiento con BI2536, como se describe en ( g ). Las imágenes de inmunofluorescencia se obtuvieron y analizaron como en la Fig. 1e. “CAROLINA DEL NORTE.” representa células sin transfección ni tratamiento con BI2536. Las barras indican medias ± SEM de análisis de no menos de 40 células con husos deformados. Prueba t de Student de dos colas para el cálculo del valor p. (i) Resumen esquemático del procedimiento experimental de (j). (j) Gráficos de dispersión de los ángulos del huso en células HeLa mitóticas transfectadas con ARNip de NDR1 más varias construcciones de NDR1 resistentes a ARNip , como se describe en (i). Las imágenes de inmunofluorescencia se obtuvieron y analizaron como en la Figura 1e. Las barras indican medias ± SEM de análisis de 40 células. Prueba t de Student de dos colas para el cálculo del valor p. Fuente: la regulación de la actividad de NDR1 por PLK1 garantiza la orientación adecuada del huso en la mitosis por Yan et al., Scientific Reports, junio de 2015.
Productos adicionales
Ensayo colorimétrico de acetilcolinesterasa Amplite®
Ensayo de peróxido de hidrógeno fluorimétrico Amplite®
Kit de cuantificación fluorimétrica de maleimida Amplite® *Fluorescencia verde*
Bibliografía
Resveratrol-induced Sirt1 phosphorylation by LKB1 mediates mitochondrial metabolism
Authors: Huang, Yuanyuan and Lu, Jianlin and Zhan, Li and Wang, Ming and Shi, Ronghua and Yuan, Xiao and Gao, Xinjiao and Liu, Xing and Zang, Jianye and Liu, Wei and others,
Journal: Journal of Biological Chemistry (2021)
Feedback control of PLK1 by Apolo1 ensures accurate chromosome segregation
Authors: Xu, Leilei and Ali, Mahboob and Duan, Wenxiu and Yuan, Xiao and Garba, Fatima and Mullen, McKay and Sun, Binwen and Poser, Ina and Duan, Hequan and Lu, Jianlin and others,
Journal: Cell Reports (2021): 109343
Methylation of PLK1 by SET7/9 ensures accurate kinetochore–microtubule dynamics
Authors: Yu, Ruoying and Wu, Huihui and Ismail, Hazrat and Du, Shihao and Cao, Jun and Wang, Jianyu and Ward, Tarsha and Yang, Fengrui and Gui, Ping and Ali, Mahboob and others,
Journal: Journal of molecular cell biology (2020): 462–476
Dynamic acetylation of the kinetochore-associated protein HEC1 ensures accurate microtubule–kinetochore attachment
Authors: Zhao, Gangyin and Cheng, Yubao and Gui, Ping and Cui, Meiying and Liu, Wei and Wang, Wenwen and Wang, Xueying and Ali, Mahboob and Dou, Zhen and Niu, Liwen and others,
Journal: Journal of Biological Chemistry (2019): 576–592
BubR1 phosphorylates CENP-E as a switch enabling the transition from lateral association to end-on capture of spindle microtubules
Authors: Huang, Yuejia and Lin, Lin and Liu, Xing and Ye, Sheng and Yao, Phil Y and Wang, Wenwen and Yang, Fengrui and Gao, Xinjiao and Li, Junying and Zhang, Yin and others,
Journal: Cell research (2019): 562–578
Methylation of PLK1 by SET7/9 ensures accurate kinetochore–microtubule dynamics
Authors: Yu, Ruoying and Wu, Huihui and Ismail, Hazrat and Du, Shihao and Cao, Jun and Wang, Jianyu and Ward, Tarsha and Yang, Fengrui and Gui, Ping and Ali, Mahboob and others, undefined
Journal: Journal of Molecular Cell Biology (2019)
ATM-Mediated Phosphorylation of Cortactin Involved in Actin Polymerization Promotes Breast Cancer Cells Migration and Invasion
Authors: Lang, Lei and Hou, Yixuan and Chen, Yanlin and Tu, Gang and Tao, Jing and Yang, Dan and Xi, Lei and Fu, Lixin and Sun, Kexin and Yin, Jiali and others, undefined
Journal: Cellular Physiology and Biochemistry (2018): 2972–2988
Discovery of Non-ATP-Competitive Inhibitors of Polo-like Kinase 1
Authors: Yun, Taikangxiang and Qin, Tan and Liu, Ying and Lai, Luhua
Journal: ChemMedChem (2016): 713–717
Cell polarity kinase MST4 cooperates with cAMP-dependent kinase to orchestrate histamine-stimulated acid secretion in gastric parietal cells
Authors: Jiang, Hao and Wang, Wenwen and Zhang, Yin and Yao, William W and Jiang, Jiying and Qin, Bo and Yao, Wendy Y and Liu, Fusheng and Wu, Huihui and Ward, Tarsha L and others, undefined
Journal: Journal of Biological Chemistry (2015): 28272–28285
Regulation of NDR1 activity by PLK1 ensures proper spindle orientation in mitosis
Authors: Yan, Maomao and Chu, Lingluo and Qin, Bo and Wang, Zhikai and Liu, Xing and Jin, Changjiang and Zhang, Guanglan and Gomez, Marta and Hergovich, Alex and er , undefined and Chen, Zhengjun and others, undefined
Journal: Scientific reports (2015): 10449
Referencias
Fluorescence detection techniques for protein kinase assay
Authors: Li Y, Xie W, Fang G.
Journal: Anal Bioanal Chem (2008): 2049
A multiplexed protein kinase assay
Authors: Shults MD, Kozlov IA, Nelson N, Kermani BG, Melnyk PC, Shevchenko V, Srinivasan A, Musmacker J, Hachmann JP, Barker DL, Lebl M, Zhao C.
Journal: Chembiochem (2007): 933
An enzyme-linked immunosorbent assay for protein kinase D activity using phosphorylation site-specific antibodies
Authors: Rykx A, Vancauwenbergh S, De Kimpe L, Janssens K, V and oninck S, Waelkens E, V and enheede JR, Van Lint J.
Journal: Assay Drug Dev Technol (2007): 637
Implementation of high-content assay for inhibitors of mitogen-activated protein kinase phosphatases
Authors: Vogt A, Lazo JS.
Journal: Methods (2007): 268
High-throughput kinase assay based on surface plasmon resonance suitable for native protein substrates
Authors: Takeda H, Fukumoto A, Miura A, Goshima N, Nomura N.
Journal: Anal Biochem (2006): 262
Assay principle for modulators of protein-protein interactions and its application to non-ATP-competitive ligands targeting protein kinase A
Authors: Saldanha SA, Kaler G, Cottam HB, Abagyan R, Taylor SS.
Journal: Anal Chem (2006): 8265
Assay development and case history of a 32K-biased library high-content MK2-EGFP translocation screen to identify p38 mitogen-activated protein kinase inhibitors on the ArrayScan 3.1 imaging platform
Authors: Trask OJ, Jr., Baker A, Williams RG, Nickischer D, K and asamy R, Laethem C, Johnston PA.
Journal: Methods Enzymol (2006): 419
A pull-down assay for 5′ AMP-activated protein kinase activity using the GST-fused protein
Authors: Kishimoto A, Ogura T, Esumi H.
Journal: Mol Biotechnol (2006): 17
A multiplexed homogeneous fluorescence-based assay for protein kinase activity in cell lysates
Authors: Shults MD, Janes KA, Lauffenburger DA, Imperiali B.
Journal: Nat Methods (2005): 277
Real-time protein kinase assay
Authors: Sun H, Low KE, Woo S, Noble RL, Graham RJ, Connaughton SS, Gee MA, Lee LG.
Journal: Anal Chem (2005): 2043
Application Notes (en Ingles)
A Library of Well-Defined and Water-Soluble Poly(alkyl phosphonate)s with Adjustable Hydrolysis
Acetylcholinesterase Inhibitory Activity of Pigment Echinochrome A
Ameliorative Effect of Novel Vitamin Formula with Herbal Extracts on Scopolamine-Induced Alzheimer’s Disease
An Increase in Plasma Homovanillic Acid with Cocoa Extract Consumption Is Associated with the Alleviation of Depressive Symptoms in Overweight or Obese Adults
Attenuation of lysyl oxidase and collagen gene expression in keratoconus patient corneal epithelium corresponds to disease severity


