
Fundamentos y principios Etiqueta His. La etiqueta de polihistidina o His6 es una etiqueta de proteína desarrollada originalmente para la purificación eficiente de proteínas en 1988 (Hochuli, Bannwarth & Döbeli et al., 1988). Como resultado de su relación costo-beneficio comparablemente grande, es conocida como una de las etiquetas de afinidad de proteínas más utilizadas hasta la fecha. Debido a su pequeño tamaño, variable entre cuatro y diez aminoácidos, rara vez interfiere con la función de la proteína objtetivo. Además, su baja inmunogenicidad y versatilidad en condiciones nativas y desnaturalizantes hacen que esta etiqueta de proteína se destaque entre otras. El principio de purificación subyacente de la etiqueta His es una interacción entre los iones metálicos y el anillo de imidazol de la histidina.
CARACTERISTICAS
Descripción general de los aspectos más importantes de la etiqueta de afinidad de proteínas
| CARACTERISTICAS DE HIS-TAG: | |||
|---|---|---|---|
| Amino Acid Sequence | HHHHHH (4-10) | ||
| DNA Sequence | 5′-CAT CAC CAT CAC CAT CAC-3′ | ||
| Size | 840.8 Da (His6) | ||
| Compatibility to recombinant proteins | Can be added either to the N- or C-Terminus of a protein | ||
| Specificity of interaction (KD) | 10 µM | ||
| Protein yield per ml of high-quality purification resin | Up to 80 mg / ml (GFP-His) | ||
| Elution conditions | Imidazole, histidine, or pH shift (pH 4-5) | ||
¿Cómo funciona una etiqueta His?
La purificación de una proteína a través del marcado con his se logra a través de una interacción entre varios iones metálicos divalentes, como Ni2+ (el ion predominantemente utilizado) y el anillo básico de imidazol de la histidina. Las etiquetas His pueden unirse a esos metales a un pH neutro o ligeramente básico (7.5 – 8 es lo típico). Vale la pena señalar que la afinidad de unión entre esos dos socios de interacción es proporcional a la longitud de la etiqueta His. Se logra una afinidad de unión diez veces mayor con las etiquetas His10 en comparación con las etiquetas His6 (Guignet, Hovius & Vogel, 2004; Fessenden, 2009). No obstante, como primera prueba, se recomienda minimizar el número de residuos de histidina para evitar posibles perturbaciones en la función de la proteína (Bornhorst & Falke, 2000).
Durante las últimas etapas del proceso de purificación, la proteína se puede eluir disminuyendo el pH a 4-5, aunque cabe señalar que algunas proteínas se desnaturalizan a un pH tan bajo. Además, la mayoría de los científicos prefieren una elución por competición con concentraciones más altas de imidazol (100-500 mM). También se recomienda insertar molaridades bajas de imidazol (5-20 mM, rara vez hasta 80 mM) en los tampones de carga y lavado para reducir los eventos de unión no específica, ya que algunas proteínas endógenas o histidinas naturales pueden mostrar una unión débil a IMAC. resinas (Bolaños-Garcia & Davies, 2006). Las condiciones óptimas para cada proteína siempre deben determinarse individualmente.
La mayoría de los protocolos de purificación por afinidad siguen los mismos tres pasos (Fig. 2):
1. Vinculación:
Se aplica a la columna una solución cruda que contiene la proteína etiquetada con His y se une en función de la interacción etiqueta de afinidad – matriz.
2. Lavado:
Otras proteínas que se unen de forma inespecífica se eliminan por lavado con buffers de lavado adecuados (ver proteína magenta en la Fig. 2). Estos buffers ya deberían contener molaridades bajas de imidazol.
3. Elución:
La proteína unida específicamente se eluye de la columna, normalmente mediante la unión competitiva de una molécula similar (p. ej., histidina e imidazol), cortando la etiqueta con una proteasa o mediante la desestabilización de la interacción etiqueta de afinidad – matriz, p. por un cambio de pH. El eluato puro se puede recolectar en tubos de reacción para futuras investigaciones.

Figura 2: proceso Bind-Wash-Elute de una purificación con etiqueta His. La concentración de imidazol (naranja) aumentó con el tiempo para eluir la proteína de interés después de realizar el lavado. Además de la elución de un paso, también es posible eluir la proteína con un gradiente. Esto es especialmente útil cuando se necesitan purezas elevadas.
Purificación de proteínas a través de His-tag
¿Cómo se purifica una proteína con una etiqueta His?
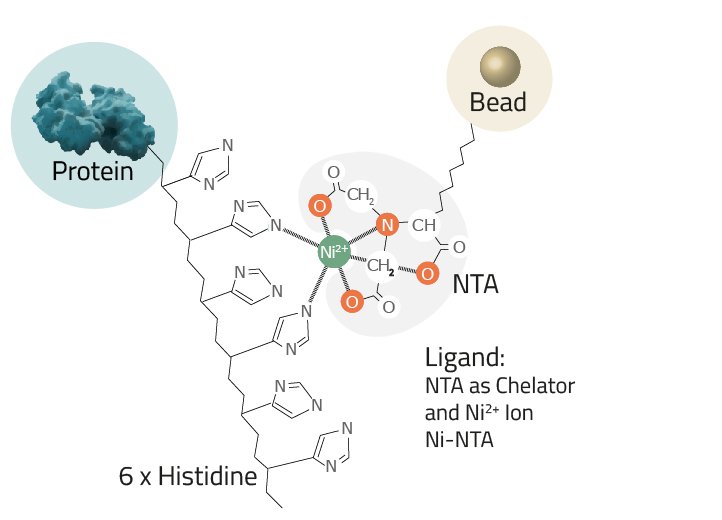
La purificación de proteínas a través de His-tag a menudo se logra mediante una cromatografía de afinidad de metal inmovilizado o, en resumen, IMAC. Este es un método de purificación ampliamente utilizado para separar proteínas y péptidos que muestran afinidad por los iones metálicos (como Ni2+ o Co2+) de una solución. El marcado con His de una proteína permite que las proteínas de fusión recién formadas se unan a los metales quelados, lo que da como resultado enriquecimientos sustanciales en muy pocos pasos. Los quelantes comunes utilizados como ligandos son las resinas de agarosa IDA y NTA. Se pueden emparejar fácilmente con un metal divalente, dependiendo de la relación de afinidad/especificidad deseada del proceso (ver Fig. 4).
Tabla 2: Comparación entre tres ligandos y sus capacidades de unión al níquel
| LIGAND | COORDINATION SITES | NICKEL BINDING |
|---|---|---|
| Iminodiacetic acid (IDA) | 3 | Weak |
| Nitrilotriacetic acid (NTA) | 4 | Moderate |
| INDIGO-Ni | 5 | Strong |
• El ligando: esta es la molécula a la que se acopla el ion metálico en un complejo quelante (ver Fig. 3).
Cube Biotech ofrece tres ligandos diferentes:
◦ NTA: El ácido nitrilotriacético es el ligando más utilizado. Su complejo quelante con níquel se muestra en la Figura 3.
◦ IDA: El ácido iminodiacético es el segundo más utilizado. Conozca las diferencias entre IDA y NTA aquí.
◦ INDIGO: Este ligando fue desarrollado aquí en Cube Biotech. Entre sus ventajas se encuentran una tolerancia al DTT y al EDTA muy aumentada. Obtenga más información sobre este nuevo ligando.
• El ion metálico acoplado: según el ion metálico acoplado al ligando, la afinidad y la especificidad de los cambios de purificación de etiquetas His. El cobre tiene la afinidad más alta, mientras que a su vez tiene la especificidad más baja. El cobalto marca el otro extremo del espectro. El níquel, el ión metálico más utilizado, tiene un buen equilibrio entre afinidad y especificidad. Regla general: cuanto mayor sea el rendimiento de proteína, menos específica se vuelve la purificación. La figura 4 puede brindarle una descripción general de la proporción de afinidad/especificidad de los iones metálicos más utilizados para la purificación de etiquetas His.
• El tamaño de las perlas: cuanto mayor sea el diámetro de las perlas utilizadas, mayor será el caudal para la purificación por FPLC. Esto los hace más útiles para medios celulares viscosos o purificación por lotes. Sin embargo, hacer uso de una velocidad de flujo más lenta generalmente aumenta el rendimiento de proteína, ya que las perlas más pequeñas tienen una mayor relación superficie-volumen. Por lo tanto, el mismo volumen de resina puede unir más proteínas que perlas de mayor tamaño. Ofrecemos diámetros de perlas de 30 µm, 40 µm, 100 µm y tamaño XL (~400 µm). Otros tamaños están disponibles bajo pedido.
Comparación de la estructura del complejo quelante y los iones metálicos:

Como añadir His-Tag
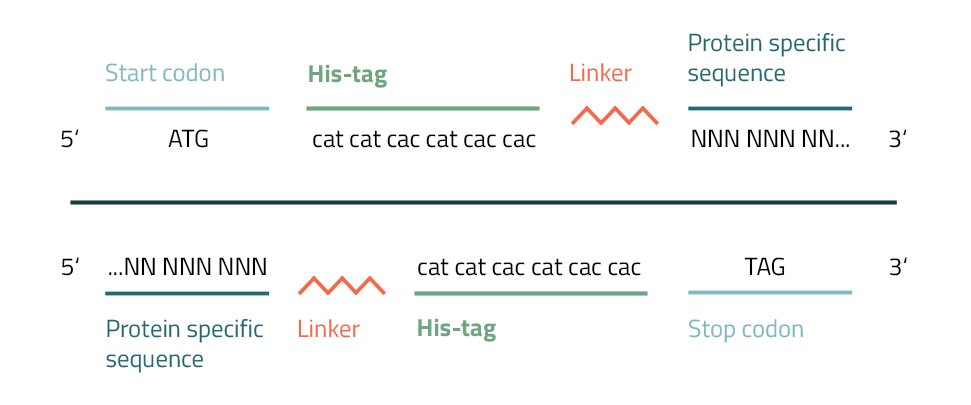
La adición de una etiqueta de afinidad de polihistidina a una proteína se puede lograr mediante varios métodos. Su pequeño tamaño y carga les permite integrarse fácilmente en los genes objetivo mediante PCR o mutagénesis dirigida al sitio.
Para lograr esto, deberá crear un cebador con codones de histidina repetitivos (CAC/CAT) junto a un codón de INICIO o DETENCIÓN. Además, se agregan a este fragmento varias bases de un extremo de su proteína de interés. La etiqueta de afinidad de hexahistidina recién incorporada agregará aproximadamente 0,9 kDa a su peso molecular. Para evitar la interferencia con la actividad de la proteína provocada por el marcado, se insertan conectores entre la etiqueta y la proteína (ver Fig. 5). Se dividen en tres grupos generales: enlazadores flexibles, enlazadores rígidos y enlazadores escindibles in vivo (Chen, Zaro & Shen, 2013). Los enlazadores comunes del grupo flexible son gly-gly-gly o gly-ser-gly. Se recomienda variar su longitud o dejarlos completamente fuera para lograr los mejores resultados para su proteína específica.
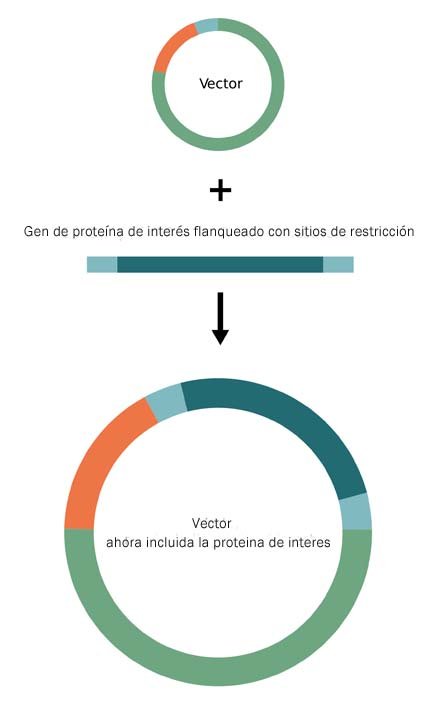
1. Vector que contiene la secuencia de polihistidina N-terminal (naranja)/sitio de clonación múltiple (azul claro)/esqueleto del vector (verde)
2. Adición de una secuencia específica de proteína (PSS) flanqueada por sitios de restricción
3. Clonación de su fragmento de código en el vector de fusión
Aplicaciones His-Tag
Además de su conocido uso como ayuda para la purificación de proteínas, las etiquetas His desempeñan un papel en muchas otras aplicaciones de la biotecnología. Ejemplos notables son los ensayos desplegables (Pulldown Assays), la asistencia en la formación de cristales o los métodos de marcaje inmunológico.
- Pulldown assays: affinity tags like His6 or GST paired with an immobilized ligand (e.g. Ni2+ agarose resin) can be used to specifically bind to a target protein complex in order to purify and identify it (Louche, Salcedo & Bigot, 2017)
- Crystal formation: an abundance of proteins can be crystallized with an intact His-tag without a negative impact on the tagged target protein (Carson et al., 2007). Smits et al. even described cases where the His-tag assisted in the formation of crystals in 2008 (Smits, Mueller & Grieshaber, 2008)
- Immune labeling: hexahistidine tags are utilized in ELISA assays, Western blot detection, flow cytometry, immunohistochemistry, or immune fluorescence
- Serial His-tagging: single subunits of a large protein can be tagged respectively to act as a marker for activity states in a gel (Howarth, Chinnapen & Gerrow et al., 2006)
- SDS-PAGE staining: metal-based fluorescence dyes facilitate the detection of a His-tagged peptide in a gel (Zhao, Hellman & Zhan et al., 2010)
- SPR immobilization strategies: surface plasmon resonance, used to determine kinetics and thermodynamics of two binding molecules, offers multiple possible immobilization strategies for protein attachment via His-Tag (Fischer, Leech, & Hubbard, 2011)
FAQ
¿Cuánto tiempo debe durar un enlazador a la etiqueta His?
La longitud del enlazador/espaciador depende de la accesibilidad estructural de la proteína diana. Las longitudes de cadena más largas tienen una mayor movilidad y, por lo tanto, son cruciales para las etiquetas con impedimento estérico (ver Fig. 7).
¿Dónde debo colocar la etiqueta His?
La etiqueta His se puede agregar al extremo N o C. La ubicación óptima es específica de la proteína y depende de la estructura molecular, ya que algunos sitios podrían perturbar la accesibilidad de la etiqueta.
¿Cómo cortar una etiqueta His?
La etiqueta de afinidad se puede eliminar mediante el uso de un sitio de escisión de proteasa insertado entre la etiqueta de polihistidina y la proteína (Nikolov, Hu & Lin et al., 1992).
¿Por qué cortar la etiqueta His?
La etiqueta His a veces se escinde para asegurar una actividad proteica intacta y una cristalización más fácil, pero aquí no se puede aplicar una regla general. Debido al tamaño y la carga relativamente pequeños, las etiquetas de His rara vez interfieren con la actividad de la proteína. Además, incluso se han descrito casos de formación de cristales asistida por etiquetas de His. Sin embargo, para aplicaciones y usos farmacéuticos, a menudo se requiere que las proteínas estén en su estado nativo, razón por la cual la escisión de la etiqueta His es un procedimiento estándar allí.
¿Cómo eluir etiquetas His de perlas?
La elución de las perlas se puede lograr con la ayuda de mayores concentraciones de imidazol, histidina o con un cambio de pH (pH 4 – 5).

Referencias
- Hochuli, E., Bannwarth, W., Döbeli, H. et al. Genetic Approach to Facilitate Purification of Recombinant Proteins with a Novel Metal Chelate Adsorbent. Nat Biotechnol 6, 1321–1325 (1988).
- Guignet, E.G., Hovius, R., Vogel, H. Reversible site-selective labeling of membrane proteins in live cells. Nature Biotechnology, 22 (4), pp. 440-444 (2004)
- Fessenden, JD., Förster resonance energy transfer measurements of ryanodine receptor type 1 structure using a novel site-specific labeling method. PLoS One, 4 (10), p. e7338 (2009)
- Bolanos-Garcia, V.M., Davies, O.R. Structural analysis and classification of native proteins from E. coli commonly co-purified by immobilised metal affinity chromatography. Biochim. Biophys. Acta, 1760, pp. 1304-1313 (2006)
- David, N.E., Gee, M., Andersen, B. et al. J Biol, Chem 272:15553 (1997).
- Skerra A. A general vector, pASK84, for cloning, bacterial production, and single-step purification of antibody Fab fragments. Gene, 141(1), 79–84 (1994).
- Petty, K J., Metal-chelate affinity chromatography. Current protocols in neuroscience vol. Chapter 5 (2001)
- Carson, M. et al., His-tag impact on structure. Acta crystallographica. Section D, Biological crystallography vol. 63, Pt 3 (2007)
- Smits, S.H., Mueller, A., Grieshaber, M.K., Schmitt, L. Coenzyme- and His-tag-induced crystallization of octopine dehydrogenase Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun., 64, pp. 836-839 (2008)
- Howarth, M., Chinnapen, DF., Gerrow, K. et al. A monovalent streptavidin with a single femtomolar biotin binding site. Nat Methods 3, 267–273 (2006)
- Chen, X., Zaro, J.L., and Shen, W.-C. Fusion protein linkers: property, design and functionality Adv. Drug Delivery Rev. 65, 1357– 1369 (2013)
- Bornhorst, J. A. & Falke, J. J. Purification of proteins using polyhistidine affinity tags. Methods in enzymology, 326, 245–254 (2000)
- Louche A., Salcedo S.P., Bigot S. Protein–Protein Interactions: Pull-Down Assays. Bacterial Protein Secretion Systems. Methods in Molecular Biology, vol 1615 (2017)
- Zhao, C., Hellman, L.M., Zhan, X., Bowman, W.S., Whiteheart, S.W., Fried, M.G. Hexahistidine-tag-specific optical probes for analyses of proteins and their interactions. Analytical Biochemistry, Vol. 399, Issue 2 (2010)
- Fischer, M., Leech, A. P. & Hubbard, R. E. Comparative assessment of different histidine-tags for immobilization of protein onto surface plasmon resonance sensorchips. Analytical chemistry, 83(5), 1800–1807 (2011)
- Nikolov, D. B., Hu, S. H., Lin, J. et al. Crystal structure of TFIID TATA-box binding protein. Nature 360, 6399 (1992)
